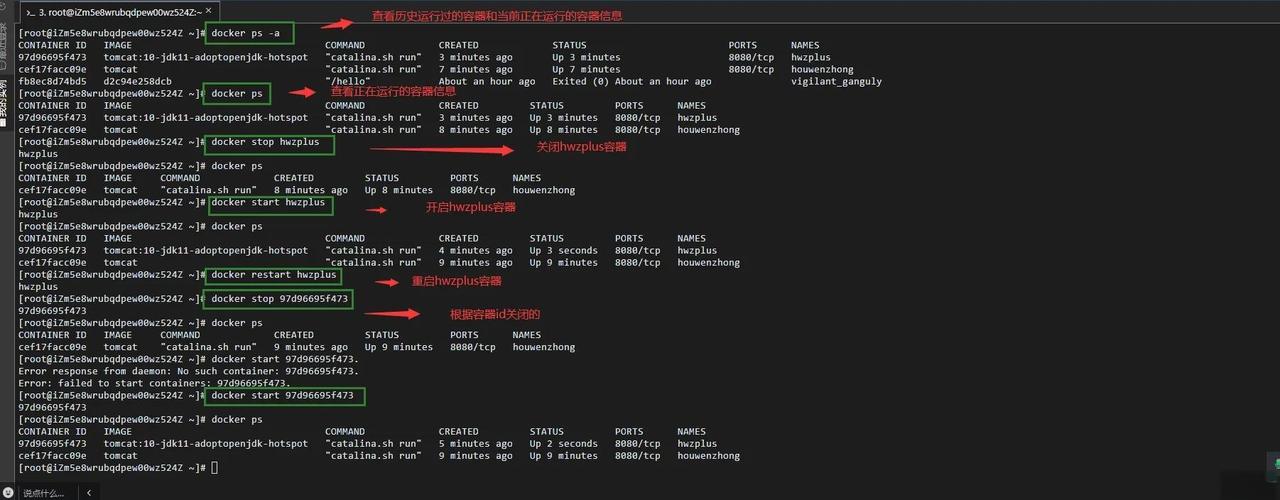
GROMACS 命令概览
GROMACS 的命令通常遵循 gmx 前缀,后面跟着具体的子命令,所有子命令都可以通过 gmx help 来查看。

基本结构:
gmx [子命令] [选项]
gmx mdrun -v -deffnm md
系统准备阶段
这个阶段的目标是将你的生物分子(如蛋白质)转换成 GROMACS 可以识别的格式,并为其构建一个合适的模拟环境。
pdb2gmx
功能: 将 PDB (Protein Data Bank) 文件转换为 GROMACS 的拓扑结构格式(.gro 和 .top)。
作用:
- 根据力场(如 CHARMM36, AMBER, OPLS-AA)为分子添加原子类型和键角参数。
- 处理缺失的原子(如蛋白质末端的氢原子)。
- 识别残基,并生成拓扑文件
topol.top。 - 生成初始坐标文件
processed.gro。
常用选项:
-f <filename.pdb>: 指定输入的 PDB 文件。-o <filename.gro>: 指定输出的坐标文件。-water <model>: 指定水模型,spc,spce,tip3p,tip4pew。-ignh: 忽略 PDB 文件中的氢原子(常用于实验结构)。-ff <forcefield>: 指定力场,charmm36,amber99sb-ildn,oplsaa。
示例:
# 使用 CHARMM36 力场和 TIP3P 水模型处理一个蛋白质 PDB 文件 gmx pdb2gmx -f protein.pdb -o processed.gro -water tip3p -ff charmm36
editconf
功能: 编辑坐标文件(.gro 或 .pdb),主要用来定义模拟盒子。
作用:
- 设置模拟盒子的形状(立方体、正交、三斜等)。
- 设置盒子的大小(边长)。
- 将分子置于盒子中心。
- 转换文件格式。
常用选项:
-f <filename.gro>: 输入坐标文件。-o <filename.gro>: 输出坐标文件。-c: 将分子置于盒子中心。-box <dx, dy, dz>: 设置立方体或正交盒子的尺寸(单位:纳米)。-d <distance>: 设置分子与盒子边界的最小距离(单位:纳米)。-bt <type>: 设置盒子类型,cubic(立方体),dodecahedron(十二面体),triclinic(三斜)。
示例:
# 将 processed.gro 中的分子放入一个距离边界至少 1.0 nm 的立方体盒子中 gmx editconf -f processed.gro -o boxed.gro -c -d 1.0 -bt cubic
genion
功能: 在已溶水的体系中添加或替换离子,以中和体系电荷或达到特定的离子浓度。 作用:
- 替换水分子为离子(如 Na+, Cl-)。
- 在体系中添加额外的离子。
常用选项:
-s <filename.tpr>: 输入一个.tpr文件(通常由gmx grompp生成)。- -o <filename.gro>`: 输出坐标文件。
- -p <filename.top>`: 输出拓扑文件(会更新)。
- -pname
`: 阳离子的名称(如 NA)。 - -nname
`: 阴离子的名称(如 CL)。 - -neutral`: 自动添加离子以中和体系总电荷。
- -conc <mol/L>`: 添加离子以达到指定的浓度。
示例:
# 首先生成输入文件 tpr gmx grompp -f ions.mdp -c boxed.gro -p topol.top -o ions.tpr # 然后使用 genion 中和体系电荷 # 注意:运行后会弹出让你选择替换哪种原子的菜单,通常选择 SOL (水) gmx genion -s ions.tpr -o ionized.gro -p topol.top -neutral
模拟运行阶段
这个阶段使用准备好的拓扑和坐标文件,通过 GROMACS 预处理程序生成二进制输入文件,然后运行动力学模拟。
gmx grompp
功能: GROMACS Preprocessor,GROMACS 模拟的编译器,它读取 .mdp (参数文件)、.top (拓扑文件) 和 .gro (坐标文件),生成一个可执行的二进制输入文件 .tpr (Portable xdr Run-input file)。
作用:
- 检查所有输入文件的兼容性。
- 将所有参数整合成一个
.tpr文件,供mdrun使用。
常用选项:
-f <filename.mdp>: 指定 MD 参数文件。-c <filename.gro>: 指定初始坐标文件。-p <filename.top>: 指定拓扑文件。-o <filename.tpr>: 指定输出的.tpr文件。-po <filename.mdp>: 输出合并了所有参数的.mdp文件(用于检查)。
示例:
# 使用 em.mdp (能量最小化参数) 文件编译能量最小化任务 gmx grompp -f em.mdp -c ionized.gro -p topol.top -o em.tpr # 使用 md.mdp (分子动力学参数) 文编译生产模拟任务 gmx grompp -f md.mdp -c ionized.gro -p topol.top -o md.tpr
gmx mdrun
功能: GROMACS 的核心执行引擎,负责运行分子动力学模拟。 作用:
- 读取
.tpr文件,执行模拟。 - 输出轨迹文件(
.xtc,.trr)、能量文件(.edr)和其他日志文件。
常用选项:
-s <filename.tpr>: 指定输入的.tpr文件。-deffnm <prefix>: 默认文件名前缀。-deffnm md会生成md.xtc,md.log,md.edr等,这是最常用的选项。-v: 详细输出,在终端显示模拟进度。- -nt
`: 使用指定的 CPU 核心数进行并行计算。 - -gpu_id
`: 指定使用的 GPU ID(如果编译时支持 GPU)。 - -cpi
从检查点文件(.cpt`)继续中断的模拟。 - -stepout
`: 每隔 N 步在屏幕和日志文件中输出一次能量信息。
示例:
# 运行一个以 md.tpr 为输入,并以 md 为前缀输出文件的模拟 gmx mdrun -s md.tpr -deffnm md -v -nt 8
模拟分析阶段
模拟结束后,需要对庞大的轨迹文件进行分析,以获得有意义的物理化学信息。
gmx energy
功能: 从 .edr 能量文件中提取特定的能量项数据,并输出为可用于绘图(如 xmgrace, gnuplot, Python/Matlab)的文本文件。
作用:
- 提取温度、压力、动能、势能、键长、角度等随时间变化的数据。
常用选项:
-f <filename.edr>: 输入能量文件。-o <filename.xvg>: 输出的 ASCII 图形文件。-b <time>: 开始分析的时间(单位:ps)。-e <time>: 结束分析的时间(单位:ps)。
示例:
# 从 md.edr 中提取温度和压力数据 # 运行后会弹出让你选择需要提取的项的菜单(输入编号即可) gmx energy -f md.edr -o temp_pressure.xvg
gmx rmsd
功能: 计算坐标均方根偏差,常用来衡量结构相对于初始构象的稳定性。 作用:
- 计算蛋白质骨架(
Backbone)或所有重原子(Heavy Atoms)的 RMSD。
常用选项:
-s <filename.tpr>: 参考结构的.tpr文件。- -f <filename.xtc>`: 轨迹文件。
- -o <filename.xvg>`: 输出的 RMSD 图形文件。
- -tu <ps|ns|fs>`: X 轴的时间单位。
示例:
# 计算蛋白质骨架相对于初始结构的 RMSD gmx rmsd -s md.tpr -f md.xtc -o rmsd.xvg -tu ns
gmx rmsf
功能: 计算坐标均方根涨落,常用来分析蛋白质的柔性区域。 作用:
- 输出每个残基或原子的 RMSF 值,残基值高则表示该区域运动剧烈。
常用选项:
-s <filename.tpr>: 参考结构的.tpr文件。- -f <filename.xtc>`: 轨迹文件。
- -o <filename.xvg>`: 输出的 RMSF 图形文件。
- -res`: 按残基输出 RMSF 值。
示例:
# 计算每个残基的 RMSF gmx rmsf -s md.tpr -f md.xtc -o rmsf.xvg -res
gmx distance
功能: 计算两个组(如两个原子、两个残基)之间的距离随时间的变化。 作用:
- 用于测量关键相互作用距离、构象变化等。
常用选项:
-s <filename.tpr>: 输入文件。- -f <filename.xtc>`: 轨迹文件。
- -o <filename.xvg>`: 输出的距离图形文件。
- -select
使用索引组选择原子(需要先用gmx make_ndx` 创建组)。
示例:
# 假设我们已经用 make_ndx 创建了名为 "res1" 和 "res2" 的组 gmx distance -s md.tpr -f md.xtc -o-dist distances.xvg -select "res1 and res2"
gmx hbond
功能: 分析氢键的形成情况。 作用:
- 计算氢键的数量、存在时间、供体-受体距离和角度等。
常用选项:
-s <filename.tpr>: 输入文件。- -f <filename.xtc>`: 轨迹文件。
- -num <filename.xvg>`: 输出氢键数量的图形文件。
- -hb <filename.hbm>`: 输出氢键详细信息。
示例:
# 分析蛋白质内部和蛋白质-水之间的氢键 gmx hbond -s md.tpr -f md.xtc -num hbond_num.xvg
gmx sasa
功能: 计算溶剂可及表面积。 作用:
- 评估分子的疏水性或暴露程度。
常用选项:
-s <filename.tpr>: 输入文件。- -f <filename.xtc>`: 轨迹文件。
- -o <filename.xvg>`: 输出的 SASA 图形文件。
示例:
# 计算蛋白质整体的溶剂可及表面积 gmx sasa -s md.tpr -f md.xtc -o sasa.xvg
gmx cluster
功能: 对轨迹进行聚类分析,找到代表性构象。 作用:
- 将模拟过程中的构象按照相似性分组,找到几个代表性的结构,用于后续分析或可视化。
常用选项:
-s <filename.tpr>: 输入文件。- -f <filename.xtc>`: 轨迹文件。
- -o <filename.xvg>`: 输出聚类大小随时间变化的图形文件。
- -cl <filename.gro>`: 输出每个簇的中心结构。
示例:
# 使用 RMSD 作为衡量标准进行聚类,找到 10 个代表性构象 gmx cluster -s md.tpr -f md.xtc -o clusters.xvg -cl cluster_structures.gro -method gromos -nstructs 10
辅助工具
gmx make_ndx
功能: 交互式地创建原子组索引。 作用:
- 这是很多分析命令(如
distance,rmsd)的前提,用于选择特定的原子集合。
使用方法:
- 运行命令:
gmx make_ndx -f md.tpr - 根据提示输入命令:
a: 选择所有原子。r 1: 选择第一个残基。name 1 Backbone: 将当前选择的组命名为 "Backbone"。q: 退出。
示例:
# 创建一个蛋白质骨架的组 gmx make_ndx -f md.tpr # (在交互界面中输入) > a C CA N O > name 1 Backbone > q
gmx trjconv
功能: 转换和处理轨迹文件。 作用:
- 改变轨迹文件的格式(
.xtc->.trr)。 - 选取轨迹的一部分(时间范围)。
- 选取体系的某一部分(如蛋白质、配体)。
- 重新设置盒子(
-pbc)。
常用选项:
-s <filename.tpr>: 输入文件(用于获取拓扑信息)。- -f <filename.xtc>`: 输入轨迹文件。
- -o <filename.xtc>`: 输出轨迹文件。
- -b
- -e
- -center`: 将选择的组置于盒子中心。
- -pbc <mol|res|atom>
处理周期性边界条件,mol(分子),res(残基),atom` (原子)。
示例:
# 选取从 10ns 到 20ns 的轨迹,并将蛋白质置于中心 gmx trjconv -s md.tpr -f md.xtc -o centered.xtc -b 10000 -e 20000 -center -pbc mol
| 阶段 | 主要命令 | 功能描述 |
|---|---|---|
| 准备 | pdb2gmx |
PDB -> GROMACS 拓扑和坐标 |
editconf |
定义模拟盒子 | |
genion |
添加/替换离子 | |
| 运行 | gmx grompp |
编译参数文件,生成 .tpr |
gmx mdrun |
执行 MD 模拟 | |
| 分析 | gmx energy |
提取能量数据 |
gmx rmsd |
计算结构偏差 | |
gmx rmsf |
计算结构涨落 | |
gmx distance |
计算原子间距离 | |
gmx hbond |
分析氢键 | |
gmx sasa |
计算溶剂可及表面积 | |
gmx cluster |
轨迹聚类分析 | |
| 辅助 | gmx make_ndx |
创建原子组索引 |
gmx trjconv |
转换和处理轨迹 |
掌握这些核心命令,你就可以完成绝大多数 GROMACS 模拟任务了,建议结合 GROMACS 官方手册和教程进行深入学习。